在建立估算器之前估算缺失值?
可以使用基本sklearn.impute.SimpleImputer將缺失值替換為平均值,中位數或最頻繁的值(眾數)。
在此示例中,我們將研究不同的插補技術:
歸因于常數0
每個特征的平均值進行歸因,并結合缺失指標輔助變量
k最近鄰歸因
迭代插補
我們將使用兩個數據集:糖尿病數據集,其中包括10個從糖尿病患者那里收集的,旨在預測疾病進展的特征變量;以及加利福尼亞住房數據集,其目標是加利福尼亞地區的平均房價。
由于這些數據集都沒有缺失值,因此我們將刪除一些值以使用人為缺失的數據創建新版本。 然后將RandomForestRegressor在完整原始數據集上的性能與使用不同技術估算的人工缺失值與變更后的數據集的性能進行比較。
print(__doc__)
# 作者: Maria Telenczuk <https://github.com/maikia>
# 執照: BSD 3 clause
下載數據并設置缺失值集
首先,我們下載兩個數據集。 scikit-learn附帶了糖尿病數據集。 它具有442個條目,每個條目具有10個功能。 加州住房數據集更大,有20640個條目和8個要素。 需要下載。 為了加快計算速度,我們將僅使用前400個條目,但可以隨意使用整個數據集。
import numpy as np
from sklearn.datasets import fetch_california_housing
from sklearn.datasets import load_diabetes
rng = np.random.RandomState(42)
X_diabetes, y_diabetes = load_diabetes(return_X_y=True)
X_california, y_california = fetch_california_housing(return_X_y=True)
X_california = X_california[:400]
y_california = y_california[:400]
def add_missing_values(X_full, y_full):
n_samples, n_features = X_full.shape
# 在75%的行中添加缺失值
missing_rate = 0.75
n_missing_samples = int(n_samples * missing_rate)
missing_samples = np.zeros(n_samples, dtype=np.bool)
missing_samples[: n_missing_samples] = True
rng.shuffle(missing_samples)
missing_features = rng.randint(0, n_features, n_missing_samples)
X_missing = X_full.copy()
X_missing[missing_samples, missing_features] = np.nan
y_missing = y_full.copy()
return X_missing, y_missing
X_miss_california, y_miss_california = add_missing_values(
X_california, y_california)
X_miss_diabetes, y_miss_diabetes = add_missing_values(
X_diabetes, y_diabetes)
插補缺失值并打分
現在,我們將編寫一個函數,該函數將對不同插補數據的結果進行評分。 讓我們分別看一下每個不良因素:
rng = np.random.RandomState(0)
from sklearn.ensemble import RandomForestRegressor
# 要使用此實驗的IterativeImputer,我們需要明確引入以下包和庫:
from sklearn.experimental import enable_iterative_imputer # noqa
from sklearn.impute import SimpleImputer, KNNImputer, IterativeImputer
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import make_pipeline
N_SPLITS = 5
regressor = RandomForestRegressor(random_state=0)
1、丟失的信息
除了估算缺失值外,注入器還具有一個add_indicator參數,該參數標記缺失的值,其中可能包含一些信息。
def get_scores_for_imputer(imputer, X_missing, y_missing):
estimator = make_pipeline(imputer, regressor)
impute_scores = cross_val_score(estimator, X_missing, y_missing,
scoring='neg_mean_squared_error',
cv=N_SPLITS)
return impute_scores
x_labels = ['Full data',
'Zero imputation',
'Mean Imputation',
'KNN Imputation',
'Iterative Imputation']
mses_california = np.zeros(5)
stds_california = np.zeros(5)
mses_diabetes = np.zeros(5)
stds_diabetes = np.zeros(5)
2、使用0替換缺失值
現在,我們將在缺失值被0代替的數據上估算分數:
def get_impute_zero_score(X_missing, y_missing):
imputer = SimpleImputer(missing_values=np.nan, add_indicator=True,
strategy='constant', fill_value=0)
zero_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return zero_impute_scores.mean(), zero_impute_scores.std()
mses_california[1], stds_california[1] = get_impute_zero_score(
X_miss_california, y_miss_california)
mses_diabetes[1], stds_diabetes[1] = get_impute_zero_score(X_miss_diabetes,
y_miss_diabetes)
3、缺失值的kNN填補
sklearn.impute.KNNImputer使用所需的最近鄰居數的加權或未加權平均值來估算缺失值。
def get_impute_knn_score(X_missing, y_missing):
imputer = KNNImputer(missing_values=np.nan, add_indicator=True)
knn_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return knn_impute_scores.mean(), knn_impute_scores.std()
mses_california[2], stds_california[2] = get_impute_knn_score(
X_miss_california, y_miss_california)
mses_diabetes[2], stds_diabetes[2] = get_impute_knn_score(X_miss_diabetes,
y_miss_diabetes)
4、均值填補缺失值
def get_impute_mean(X_missing, y_missing):
imputer = SimpleImputer(missing_values=np.nan, strategy="mean",
add_indicator=True)
mean_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return mean_impute_scores.mean(), mean_impute_scores.std()
mses_california[3], stds_california[3] = get_impute_mean(X_miss_california,
y_miss_california)
mses_diabetes[3], stds_diabetes[3] = get_impute_mean(X_miss_diabetes,
y_miss_diabetes)
5、缺失值的迭代估算
另一個選項是sklearn.impute.IterativeImputer。 這使用循環線性回歸,依次將缺少值的每個特征建模為其他特征的函數。 所實現的版本假定為高斯(輸出)變量。 如果您的功能顯然不正常,請考慮將其轉換為看起來更正常,以潛在地提高性能。
def get_impute_iterative(X_missing, y_missing):
imputer = IterativeImputer(missing_values=np.nan, add_indicator=True,
random_state=0, n_nearest_features=5,
sample_posterior=True)
iterative_impute_scores = get_scores_for_imputer(imputer,
X_missing,
y_missing)
return iterative_impute_scores.mean(), iterative_impute_scores.std()
mses_california[4], stds_california[4] = get_impute_iterative(
X_miss_california, y_miss_california)
mses_diabetes[4], stds_diabetes[4] = get_impute_iterative(X_miss_diabetes,
y_miss_diabetes)
mses_diabetes = mses_diabetes * -1
mses_california = mses_california * -1
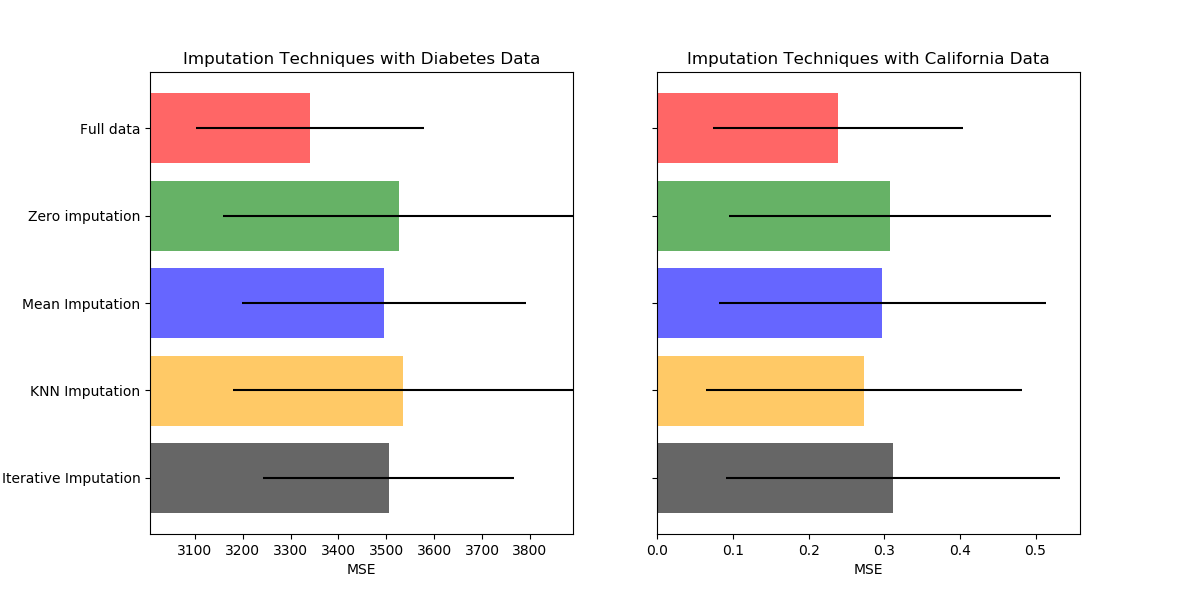
繪制結果
最后,我們將可視化分數計算并輸出:
import matplotlib.pyplot as plt
n_bars = len(mses_diabetes)
xval = np.arange(n_bars)
colors = ['r', 'g', 'b', 'orange', 'black']
# 繪制糖尿病數據集的結果
plt.figure(figsize=(12, 6))
ax1 = plt.subplot(121)
for j in xval:
ax1.barh(j, mses_diabetes[j], xerr=stds_diabetes[j],
color=colors[j], alpha=0.6, align='center')
ax1.set_title('Imputation Techniques with Diabetes Data')
ax1.set_xlim(left=np.min(mses_diabetes) * 0.9,
right=np.max(mses_diabetes) * 1.1)
ax1.set_yticks(xval)
ax1.set_xlabel('MSE')
ax1.invert_yaxis()
ax1.set_yticklabels(x_labels)
# 繪制加利福尼亞房屋價值數據集的結果
ax2 = plt.subplot(122)
for j in xval:
ax2.barh(j, mses_california[j], xerr=stds_california[j],
color=colors[j], alpha=0.6, align='center')
ax2.set_title('Imputation Techniques with California Data')
ax2.set_yticks(xval)
ax2.set_xlabel('MSE')
ax2.invert_yaxis()
ax2.set_yticklabels([''] * n_bars)
plt.show()
# 你也可以嘗試其他技巧。例如,對于具有高強度變量的數據,
# 中位數是一個更可靠的估計器,該變量可能會主導結果(否則會出現長尾現象)。
輸出:
腳本的總運行時間:0分鐘18.774秒
