具有多重共線性或相關特征的置換重要性?
在本例中,我們使用 permutation_importance計算 Wisconsin乳腺癌數據集上的置換重要性。 RandomForestClassifier可以很容易地在測試數據集上獲得97%的準確率。因為這個數據集包含多重共性特征,所以置換的重要性將顯示沒有一個特征是重要的。處理多重共線性的一種方法是對特征的Spearman秩序相關性進行分層聚類,選擇一個閾值,并從每個聚類中保留一個特征。
Note: See also Permutation Importance vs Random Forest Feature Importance (MDI)
print(__doc__)
from collections import defaultdict
import matplotlib.pyplot as plt
import numpy as np
from scipy.stats import spearmanr
from scipy.cluster import hierarchy
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import permutation_importance
from sklearn.model_selection import train_test_split
乳腺癌數據上的隨機森林特征重要性
首先,我們在乳腺癌數據集上訓練隨機森林,并在測試集上評估其準確性:
data = load_breast_cancer()
X, y = data.data, data.target
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
print("Accuracy on test data: {:.2f}".format(clf.score(X_test, y_test)))
Accuracy on test data: 0.97
其次,我們繪制了基于樹的特征重要性和置換重要性。置換重要性圖顯示,變換一個特征最多降低了0.012的準確性,這意味著沒有任何一個特征是重要的。這與上面計算的高精度測試是矛盾的:某些特性必須是重要的。在訓練集上計算置換的重要性,以顯示模型在訓練過程中對每個特征的依賴程度。
result = permutation_importance(clf, X_train, y_train, n_repeats=10,
random_state=42)
perm_sorted_idx = result.importances_mean.argsort()
tree_importance_sorted_idx = np.argsort(clf.feature_importances_)
tree_indices = np.arange(0, len(clf.feature_importances_)) + 0.5
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
ax1.barh(tree_indices,
clf.feature_importances_[tree_importance_sorted_idx], height=0.7)
ax1.set_yticklabels(data.feature_names[tree_importance_sorted_idx])
ax1.set_yticks(tree_indices)
ax1.set_ylim((0, len(clf.feature_importances_)))
ax2.boxplot(result.importances[perm_sorted_idx].T, vert=False,
labels=data.feature_names[perm_sorted_idx])
fig.tight_layout()
plt.show()
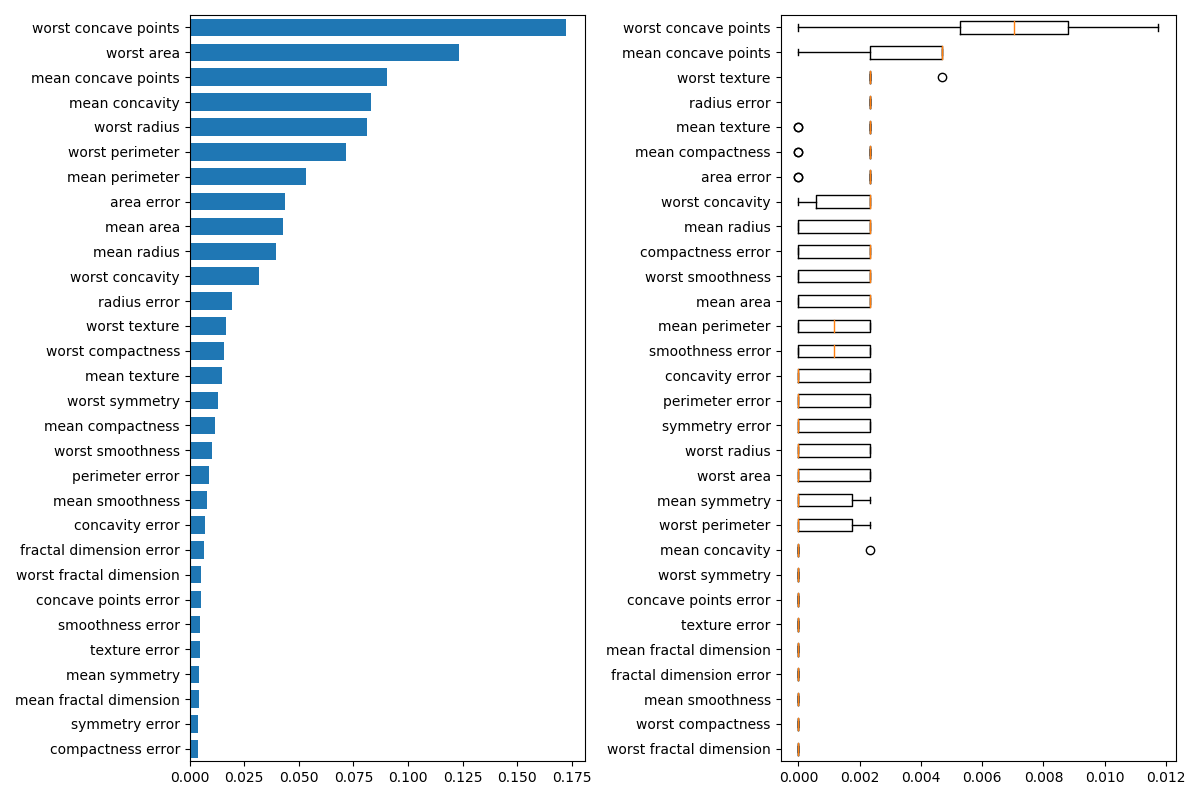
處理多重共線性特征
當特征是共線特征時,置換一個特征對模型性能的影響很小,因為它可以從相關特征中獲得相同的信息。
處理多線性特征的一種方法是對Spearman秩序相關性執行分層聚類,選擇一個閾值,并從每個聚類中保留一個特征。首先,我們繪制了相關特征的熱圖:
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
corr = spearmanr(X).correlation
corr_linkage = hierarchy.ward(corr)
dendro = hierarchy.dendrogram(corr_linkage, labels=data.feature_names, ax=ax1,
leaf_rotation=90)
dendro_idx = np.arange(0, len(dendro['ivl']))
ax2.imshow(corr[dendro['leaves'], :][:, dendro['leaves']])
ax2.set_xticks(dendro_idx)
ax2.set_yticks(dendro_idx)
ax2.set_xticklabels(dendro['ivl'], rotation='vertical')
ax2.set_yticklabels(dendro['ivl'])
fig.tight_layout()
plt.show()
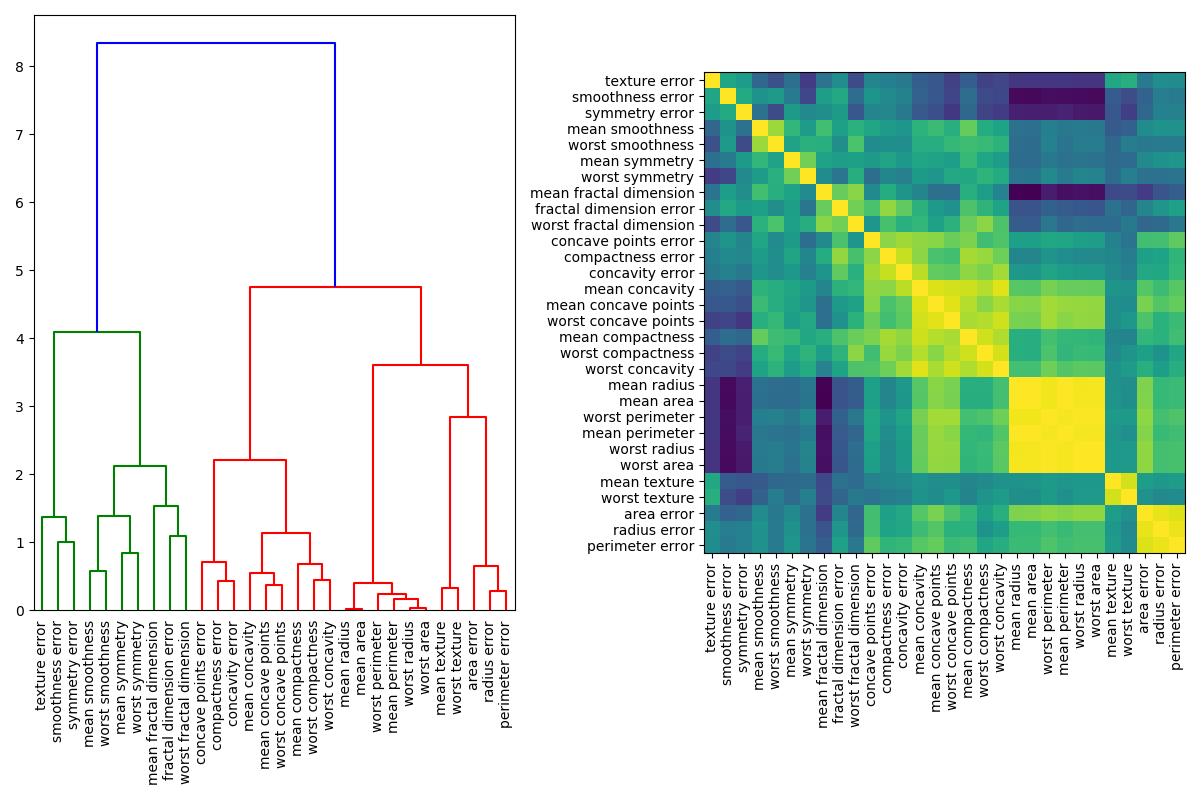 接下來,我們通過對樹狀圖的可視化檢查來手動選擇一個閾值,將我們的特征分組成簇,并從每個簇中選擇一個特征來保存,從我們的數據集中選擇這些特征,并訓練一個新的隨機森林。與在完整數據集上訓練的隨機林相比,新隨機林的測試精度變化不大。
接下來,我們通過對樹狀圖的可視化檢查來手動選擇一個閾值,將我們的特征分組成簇,并從每個簇中選擇一個特征來保存,從我們的數據集中選擇這些特征,并訓練一個新的隨機森林。與在完整數據集上訓練的隨機林相比,新隨機林的測試精度變化不大。
Accuracy on test data with features removed: 0.97
腳本的總運行時間:(0分6.505秒)
Download Python source code:plot_permutation_importance_multicollinear.py
Download Jupyter notebook:plot_permutation_importance_multicollinear.ipynb
