Johnson-Lindenstrauss綁定嵌入隨機投影?
Johnson-Lindenstrauss引理指出,任何高維數據集都可以隨機投影到低維歐幾里得空間中,同時控制成對距離的畸變。
print(__doc__)
import sys
from time import time
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from distutils.version import LooseVersion
from sklearn.random_projection import johnson_lindenstrauss_min_dim
from sklearn.random_projection import SparseRandomProjection
from sklearn.datasets import fetch_20newsgroups_vectorized
from sklearn.datasets import load_digits
from sklearn.metrics.pairwise import euclidean_distances
# 不推薦使用“規范”選項,更贊成使用直方圖中的“密度”
if LooseVersion(matplotlib.__version__) >= '2.1':
density_param = {'density': True}
else:
density_param = {'normed': True}
理論界限
隨機投影p引入的失真是由以下事實所斷定的:p正在以很高的概率定義eps嵌入,其定義如下:
其中u和v是從形狀[n_samples,n_features]形狀的數據集中獲取的任何行,而p是形狀為[n_components,n_features]的隨機高斯N(0,1)矩陣(或稀疏的Achlioptas矩陣)的投影。
保證eps嵌入的最小組件數由下式給出:
第一個圖顯示,隨著樣本數量n_samples的增加,最小維度n_components的數量對數地增加,以保證eps嵌入。
# 允許變形的范圍
eps_range = np.linspace(0.1, 0.99, 5)
colors = plt.cm.Blues(np.linspace(0.3, 1.0, len(eps_range)))
# 嵌入的樣本數(觀測值)范圍
n_samples_range = np.logspace(1, 9, 9)
plt.figure()
for eps, color in zip(eps_range, colors):
min_n_components = johnson_lindenstrauss_min_dim(n_samples_range, eps=eps)
plt.loglog(n_samples_range, min_n_components, color=color)
plt.legend(["eps = %0.1f" % eps for eps in eps_range], loc="lower right")
plt.xlabel("Number of observations to eps-embed")
plt.ylabel("Minimum number of dimensions")
plt.title("Johnson-Lindenstrauss bounds:\nn_samples vs n_components")
plt.show()
輸出:
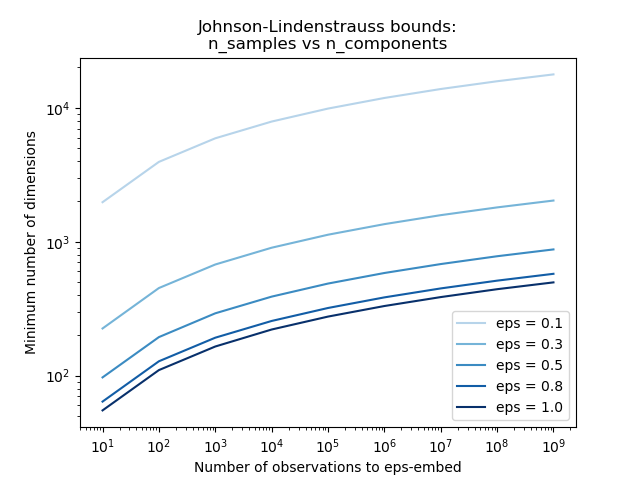
第二個圖顯示,對于給定數量的樣本n_samples,允許失真eps的增加允許大幅減少最小維數n_components:
# 允許變形的范圍
eps_range = np.linspace(0.01, 0.99, 100)
# 嵌入的樣本數(觀測值)范圍
n_samples_range = np.logspace(2, 6, 5)
colors = plt.cm.Blues(np.linspace(0.3, 1.0, len(n_samples_range)))
plt.figure()
for n_samples, color in zip(n_samples_range, colors):
min_n_components = johnson_lindenstrauss_min_dim(n_samples, eps=eps_range)
plt.semilogy(eps_range, min_n_components, color=color)
plt.legend(["n_samples = %d" % n for n in n_samples_range], loc="upper right")
plt.xlabel("Distortion eps")
plt.ylabel("Minimum number of dimensions")
plt.title("Johnson-Lindenstrauss bounds:\nn_components vs eps")
plt.show()
輸出:
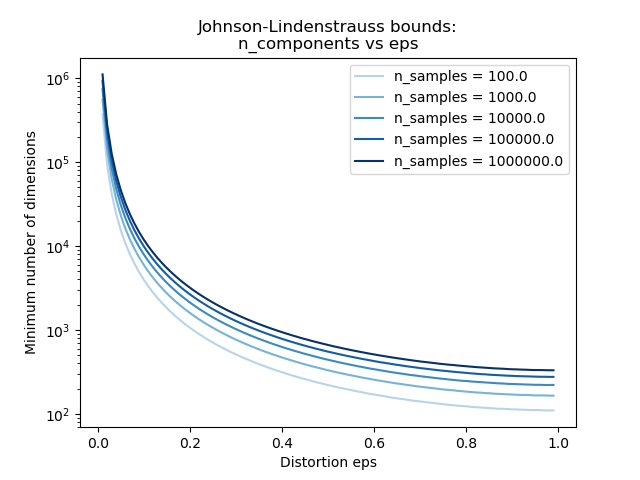
實證驗證
我們在20個新聞組文本文檔(TF-IDF詞頻)數據集或數字數據集上驗證上述界限:
對于20個新聞組數據集,使用稀疏隨機矩陣將總共500個具有100k要素的文檔投影到較小的歐幾里得空間,并為目標n_components維數提供不同的值。
對于數字數據集,用于500個手寫數字圖片的一些8x8灰度像素數據被隨機投影到空間,用于各種較大數量的維n_component。
默認數據集是20個新聞組數據集。 要在digits數據集上運行示例,請將--use-digits-dataset命令行參數傳遞給此腳本。
if '--use-digits-dataset' in sys.argv:
data = load_digits().data[:500]
else:
data = fetch_20newsgroups_vectorized().data[:500]
對于n_components的每個值,我們繪制:
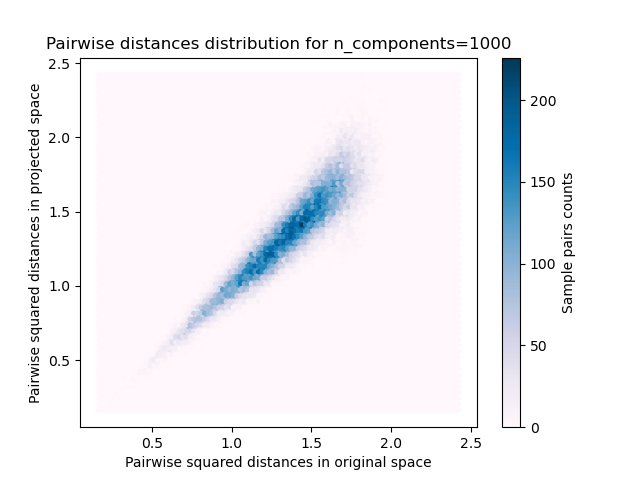
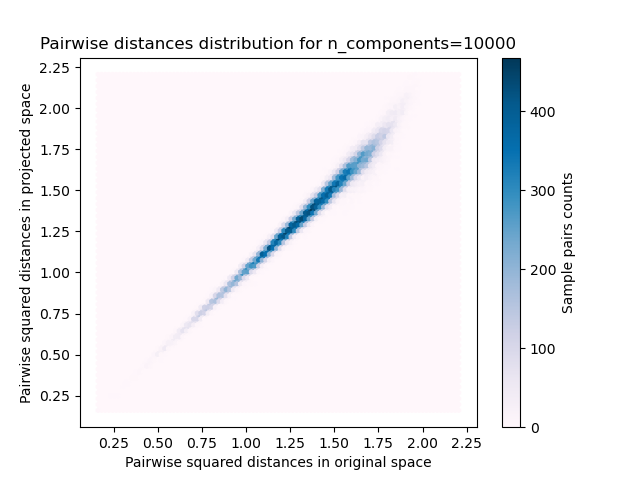
原始和投影空間中成對距離的樣本對的二維分布分別為x和y軸。
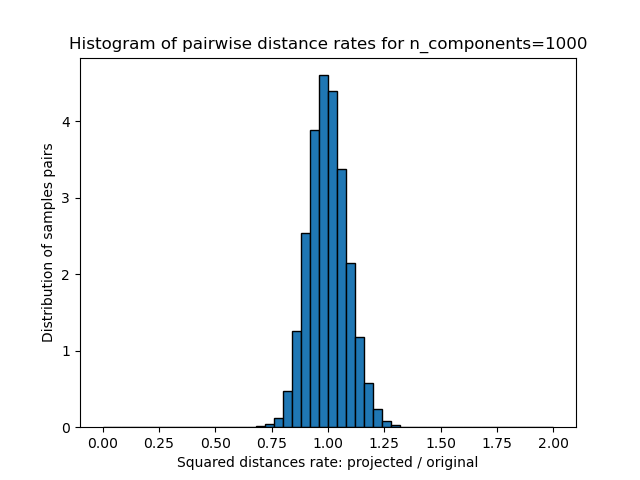
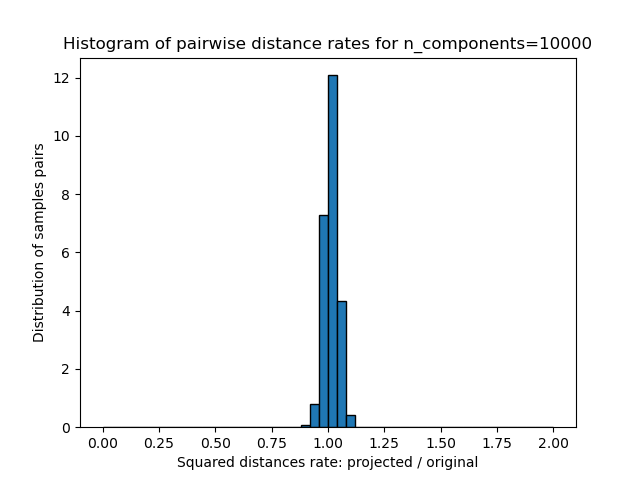
這些距離的比例的一維直方圖(投影/原始)。
n_samples, n_features = data.shape
print("Embedding %d samples with dim %d using various random projections"
% (n_samples, n_features))
n_components_range = np.array([300, 1000, 10000])
dists = euclidean_distances(data, squared=True).ravel()
# 僅選擇不相同的樣本對
nonzero = dists != 0
dists = dists[nonzero]
for n_components in n_components_range:
t0 = time()
rp = SparseRandomProjection(n_components=n_components)
projected_data = rp.fit_transform(data)
print("Projected %d samples from %d to %d in %0.3fs"
% (n_samples, n_features, n_components, time() - t0))
if hasattr(rp, 'components_'):
n_bytes = rp.components_.data.nbytes
n_bytes += rp.components_.indices.nbytes
print("Random matrix with size: %0.3fMB" % (n_bytes / 1e6))
projected_dists = euclidean_distances(
projected_data, squared=True).ravel()[nonzero]
plt.figure()
min_dist = min(projected_dists.min(), dists.min())
max_dist = max(projected_dists.max(), dists.max())
plt.hexbin(dists, projected_dists, gridsize=100, cmap=plt.cm.PuBu,
extent=[min_dist, max_dist, min_dist, max_dist])
plt.xlabel("Pairwise squared distances in original space")
plt.ylabel("Pairwise squared distances in projected space")
plt.title("Pairwise distances distribution for n_components=%d" %
n_components)
cb = plt.colorbar()
cb.set_label('Sample pairs counts')
rates = projected_dists / dists
print("Mean distances rate: %0.2f (%0.2f)"
% (np.mean(rates), np.std(rates)))
plt.figure()
plt.hist(rates, bins=50, range=(0., 2.), edgecolor='k', **density_param)
plt.xlabel("Squared distances rate: projected / original")
plt.ylabel("Distribution of samples pairs")
plt.title("Histogram of pairwise distance rates for n_components=%d" %
n_components)
# 待完成:計算eps的期望值并將其添加到上一個圖
# 作為垂直線/區域
plt.show()
輸出:
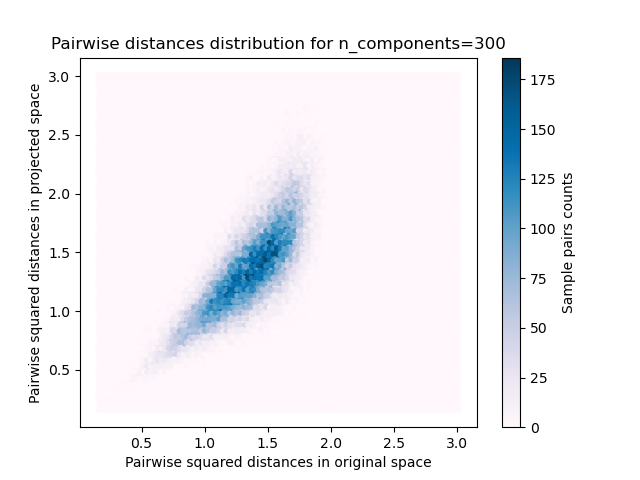
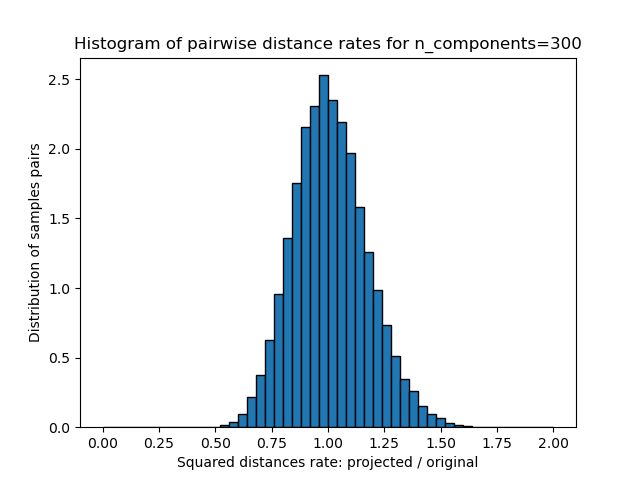
Embedding 500 samples with dim 130107 using various random projections
Projected 500 samples from 130107 to 300 in 0.969s
Random matrix with size: 1.303MB
Mean distances rate: 1.01 (0.16)
Projected 500 samples from 130107 to 1000 in 3.381s
Random matrix with size: 4.325MB
Mean distances rate: 1.04 (0.11)
Projected 500 samples from 130107 to 10000 in 33.519s
Random matrix with size: 43.285MB
Mean distances rate: 0.99 (0.03)
我們可以看到,對于n_component的低值,分布較寬,具有許多扭曲對和偏斜的分布(由于左側的零比率的硬性限制,因為距離始終為正),而對于n_component的較大值,則可以控制失真,并且通過隨機投影可以很好地保持距離。
備注
根據JL引理,投影500個樣本而沒有太多失真將需要至少數千個維度,而與原始數據集的特征數量無關。
因此,對在輸入空間中僅具有64個特征的數字數據集使用隨機投影是沒有意義的:在這種情況下,它不允許降維。
另一方面,在二十個新聞組中,維數可以從56436降低到10000,同時合理地保持成對的距離。
腳本的總運行時間:(0分鐘43.639秒)
