RBF內核的顯式特征圖近似?
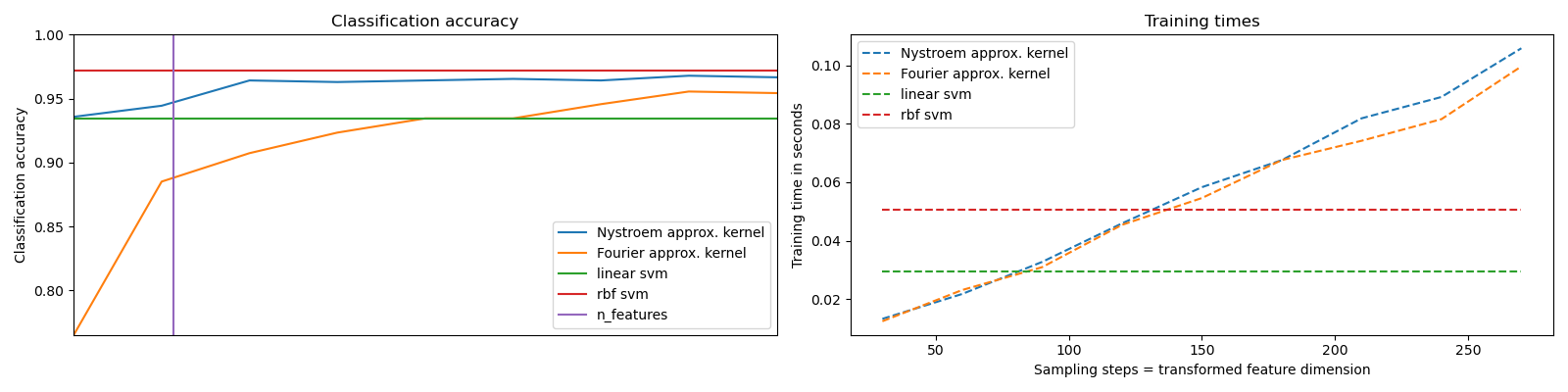
一個示例,說明了RBF內核的特征圖的近似值。
它顯示了如何使用RBFSampler和Nystroem來近似RBF內核的特征圖,以便在數字數據集上使用SVM進行分類。比較了使用原始空間中的線性SVM,使用近似映射的線性SVM和使用內核化SVM的結果。顯示了不同數量的蒙特卡洛采樣的時間和精度(在RBFSampler的情況下,它使用隨機傅里葉特征)和訓練集的不同大小的子集(對于Nystroem)用于近似映射。
請注意,這里的數據集不足以顯示核近似的好處,因為精確的SVM仍然相當快。
對更多維度進行采樣顯然會帶來更好的分類結果,但代價更高。這意味著在運行時間和精度之間需要權衡,這由參數n_components給出。請注意,通過sklearn.linear_model.SGDClassifier使用隨機梯度下降可大大加快求解線性SVM以及近似內核SVM的速度。對于內核化SVM,這是不容易實現的。
Python包和數據集導入,加載數據集
# 作者: Gael Varoquaux <gael dot varoquaux at normalesup dot org>
# Andreas Mueller <amueller@ais.uni-bonn.de>
# 執照: BSD 3 clause
print(__doc__)
# 標準的、科學的Python包導入過程
import matplotlib.pyplot as plt
import numpy as np
from time import time
# 導入數據集,分類器和性能指標
from sklearn import datasets, svm, pipeline
from sklearn.kernel_approximation import (RBFSampler,
Nystroem)
from sklearn.decomposition import PCA
# 手寫數字數據集
digits = datasets.load_digits(n_class=9)
時序圖和精度圖
要將分類器應用于此數據,我們需要將圖像展平,以將數據轉換為(樣本,特征)矩陣:
n_samples = len(digits.data)
data = digits.data / 16.
data -= data.mean(axis=0)
# 對手寫數字數據集的前半段進行學習
data_train, targets_train = (data[:n_samples // 2],
digits.target[:n_samples // 2])
# 預測手寫數字數據集的后半段
data_test, targets_test = (data[n_samples // 2:],
digits.target[n_samples // 2:])
# data_test = scaler.transform(data_test)
# 創造分類器:一個支持向量機
kernel_svm = svm.SVC(gamma=.2)
linear_svm = svm.LinearSVC()
# 用核估計方式創建管道
# 和線性svm
feature_map_fourier = RBFSampler(gamma=.2, random_state=1)
feature_map_nystroem = Nystroem(gamma=.2, random_state=1)
fourier_approx_svm = pipeline.Pipeline([("feature_map", feature_map_fourier),
("svm", svm.LinearSVC())])
nystroem_approx_svm = pipeline.Pipeline([("feature_map", feature_map_nystroem),
("svm", svm.LinearSVC())])
# 用線性及帶核的支持向量機進行擬合和預測:
kernel_svm_time = time()
kernel_svm.fit(data_train, targets_train)
kernel_svm_score = kernel_svm.score(data_test, targets_test)
kernel_svm_time = time() - kernel_svm_time
linear_svm_time = time()
linear_svm.fit(data_train, targets_train)
linear_svm_score = linear_svm.score(data_test, targets_test)
linear_svm_time = time() - linear_svm_time
sample_sizes = 30 * np.arange(1, 10)
fourier_scores = []
nystroem_scores = []
fourier_times = []
nystroem_times = []
for D in sample_sizes:
fourier_approx_svm.set_params(feature_map__n_components=D)
nystroem_approx_svm.set_params(feature_map__n_components=D)
start = time()
nystroem_approx_svm.fit(data_train, targets_train)
nystroem_times.append(time() - start)
start = time()
fourier_approx_svm.fit(data_train, targets_train)
fourier_times.append(time() - start)
fourier_score = fourier_approx_svm.score(data_test, targets_test)
nystroem_score = nystroem_approx_svm.score(data_test, targets_test)
nystroem_scores.append(nystroem_score)
fourier_scores.append(fourier_score)
# 繪制結果:
plt.figure(figsize=(16, 4))
accuracy = plt.subplot(121)
# 設置次坐標軸,顯示時間
timescale = plt.subplot(122)
accuracy.plot(sample_sizes, nystroem_scores, label="Nystroem approx. kernel")
timescale.plot(sample_sizes, nystroem_times, '--',
label='Nystroem approx. kernel')
accuracy.plot(sample_sizes, fourier_scores, label="Fourier approx. kernel")
timescale.plot(sample_sizes, fourier_times, '--',
label='Fourier approx. kernel')
# 繪制rbf和線性核的橫線
accuracy.plot([sample_sizes[0], sample_sizes[-1]],
[linear_svm_score, linear_svm_score], label="linear svm")
timescale.plot([sample_sizes[0], sample_sizes[-1]],
[linear_svm_time, linear_svm_time], '--', label='linear svm')
accuracy.plot([sample_sizes[0], sample_sizes[-1]],
[kernel_svm_score, kernel_svm_score], label="rbf svm")
timescale.plot([sample_sizes[0], sample_sizes[-1]],
[kernel_svm_time, kernel_svm_time], '--', label='rbf svm')
# 為數據集的維度設置豎線
accuracy.plot([64, 64], [0.7, 1], label="n_features")
# 圖例和標題
accuracy.set_title("Classification accuracy")
timescale.set_title("Training times")
accuracy.set_xlim(sample_sizes[0], sample_sizes[-1])
accuracy.set_xticks(())
accuracy.set_ylim(np.min(fourier_scores), 1)
timescale.set_xlabel("Sampling steps = transformed feature dimension")
accuracy.set_ylabel("Classification accuracy")
timescale.set_ylabel("Training time in seconds")
accuracy.legend(loc='best')
timescale.legend(loc='best')
plt.tight_layout()
plt.show()
輸出:
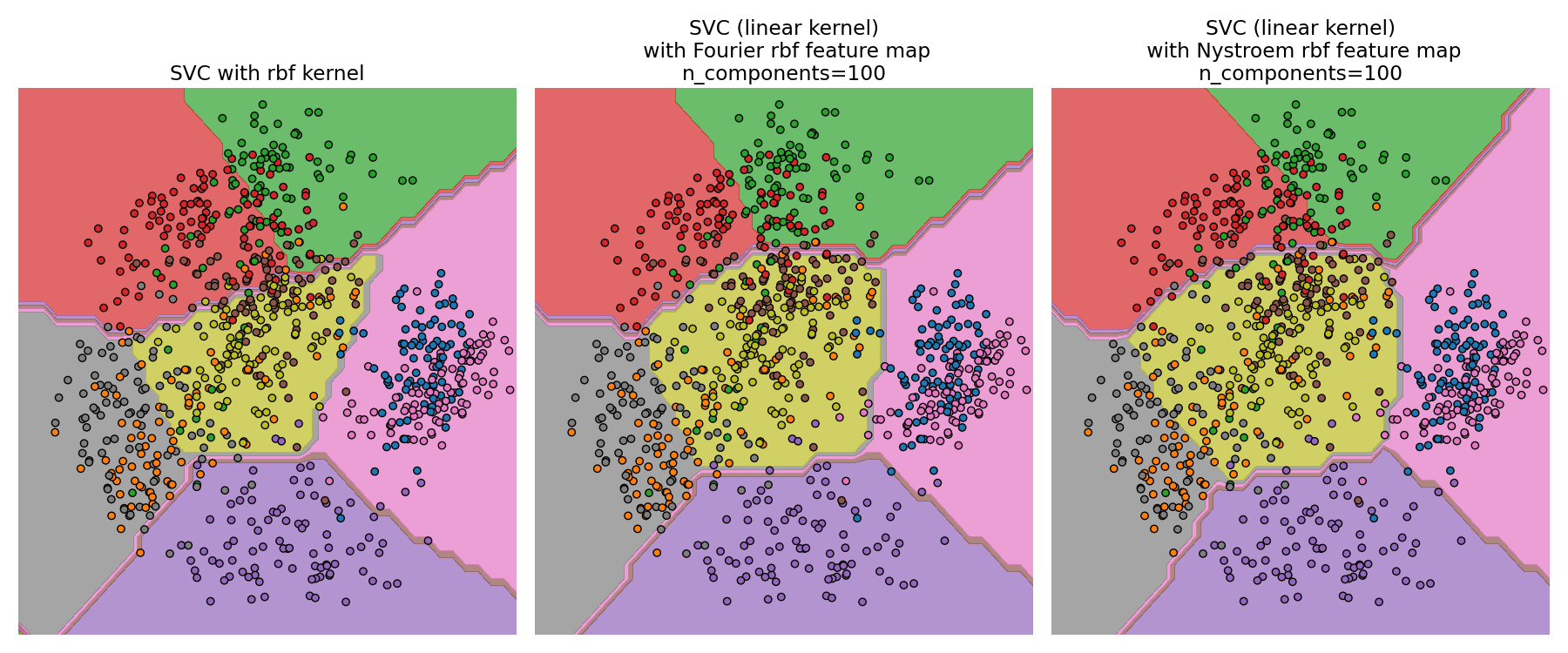
RBF內核SVM和線性SVM的決策平面
第二個圖顯示了帶有近似內核圖的RBF內核SVM和線性SVM的決策平面。該圖顯示了投影到數據的前兩個主要成分上的分類器的決策面。 這種可視化應該不費吹灰之力就能完成,因為它只是決策面上64個維度中的一個有趣切片。 特別要注意的是,數據點(用點表示)不一定要分類到它所在的區域,因為它不會位于前兩個主要成分所跨越的平面上。 內核近似中詳細介紹了RBFSampler和Nystroem的用法。
# 可視化決策平面,向下投影到數據集的前兩個主要組成部分
pca = PCA(n_components=8).fit(data_train)
X = pca.transform(data_train)
# 沿著前兩個主要成分生成網格
multiples = np.arange(-2, 2, 0.1)
# 沿第一成分走
first = multiples[:, np.newaxis] * pca.components_[0, :]
# 沿第二成分走
second = multiples[:, np.newaxis] * pca.components_[1, :]
# 結合起來
grid = first[np.newaxis, :, :] + second[:, np.newaxis, :]
flat_grid = grid.reshape(-1, data.shape[1])
# 為圖像設置標題
titles = ['SVC with rbf kernel',
'SVC (linear kernel)\n with Fourier rbf feature map\n'
'n_components=100',
'SVC (linear kernel)\n with Nystroem rbf feature map\n'
'n_components=100']
plt.figure(figsize=(18, 7.5))
plt.rcParams.update({'font.size': 14})
# 預測并繪圖
for i, clf in enumerate((kernel_svm, nystroem_approx_svm,
fourier_approx_svm)):
# 繪制決策邊界
# 為此,我們將為網格[x_min,x_max] x [y_min,y_max]中的每個點分配顏色
plt.subplot(1, 3, i + 1)
Z = clf.predict(flat_grid)
# 將結果放入彩色的繪圖
Z = Z.reshape(grid.shape[:-1])
plt.contourf(multiples, multiples, Z, cmap=plt.cm.Paired)
plt.axis('off')
# 同時,繪制訓練數據點
plt.scatter(X[:, 0], X[:, 1], c=targets_train, cmap=plt.cm.Paired,
edgecolors=(0, 0, 0))
plt.title(titles[i])
plt.tight_layout()
plt.show()
輸出:
腳本的總運行時間:(0分鐘2.195秒)
